生命科學與技術學院張赫團隊關於染色質拓撲相關結構域調控基因轉錄新模式的最新研究成果發表於《基因組蛋白質組與生物信息學報》
來源:生命科學與技術學院
時間👨🏻🦰:2024-05-20 瀏覽:
真核生物基因組在進化上保守,但在空間層次上具有高度可塑性,由染色質環、拓撲相關結構域(topologically associating domain9️⃣,TAD)等動態的染色質高維結構組成。當TAD重構介導的染色質環處於紊亂狀態時,易引起染色質構象重塑💆♂️,導致基因表達異常,甚至促使腫瘤發生。因此💋,亟需進一步發現受TAD變化調控的功能基因☝🏿,並深入揭示TAD重構調控基因轉錄的新機製。
近日✍🏻,生命科學與技術學院張赫團隊在《基因組蛋白質組與生物信息學報》(Genomics Proteomics Bioinformatics)上發表了題為“Inter3D: Capture of TAD Reorganization Endows Variant Patterns of Gene Transcription”的最新研究成果,首次建立了一種名為Inter3D的新方法,利用該方法特異性地識別TAD重構介導的功能染色質環,揭示基因轉錄調控新模式。
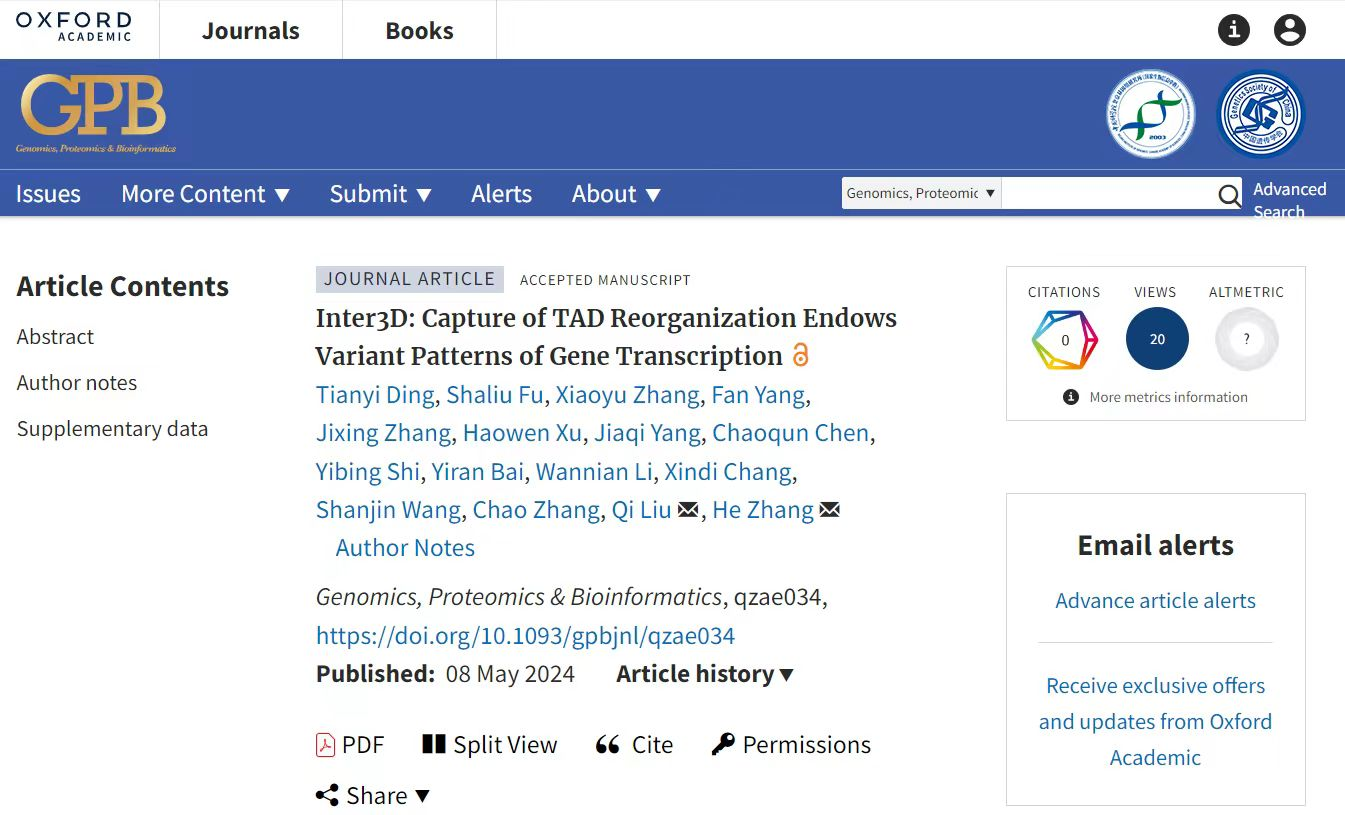
Inter3D分析方法用於分析順式轉錄調控元件(cis regulatory element🔅,CRE)與基因啟動子間的相互作用🤹♀️🦹🏿♀️。TAD重構存在兩種不同機製,即Intra-TAD(Mechanism 1👸🏽,CTCF促進啟動子–CRE相互作用)或Inter-TAD(Mechanism 2,CTCF阻礙啟動子–CRE相互作用),在調控基因轉錄方面發揮著關鍵作用。Inter3D首先使用Hi-C和CTCF ChIP-seq數據集來識別CTCF介導的TAD邊界變化,並結合RNA-seq數據集識別出由CTCF結合變化和TAD重構調控的顯著差異表達基因(differentially expressed genes,DEGs),再通過ATAC-seq數據集的疊加🥞,在啟動子遠端的峰值中識別候選增強子🫳🏿🛥,排除僅受三維結構而非增強子調控的假陽性靶基因。通過Inter3D方法🌴,該研究成功計算出基於Inter-TAD(Mechanism 1)的8個癌基因和28個抑製基因,以及基於Intra-TAD(Mechanism 2)的11個癌基因和26個抑製基因。
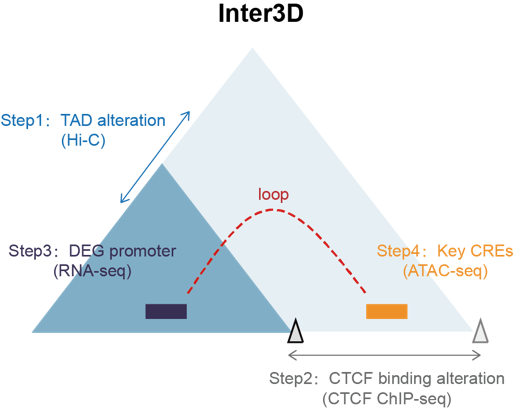
為了進一步檢測Inter3D方法的準確性🏋🏼,該研究挑選了抑癌基因MYL12B和促癌基因CYP27B1。40 Kb分辨率結果顯示,相較於正常細胞ARPE19⚪️⬛️,在腫瘤細胞WERI-RB1中,18號染色體上的MYL12B基因座處存在一個新生TAD(neo-TAD)邊界🧑🏼🚒,發生TAD分離現象🎅🏼,導致MYL12B啟動子(P1)與其下遊增強子(E1)之間染色質環特異性丟失,引起WERI-RB1細胞中MYL12B轉錄水平顯著降低👵🏽。
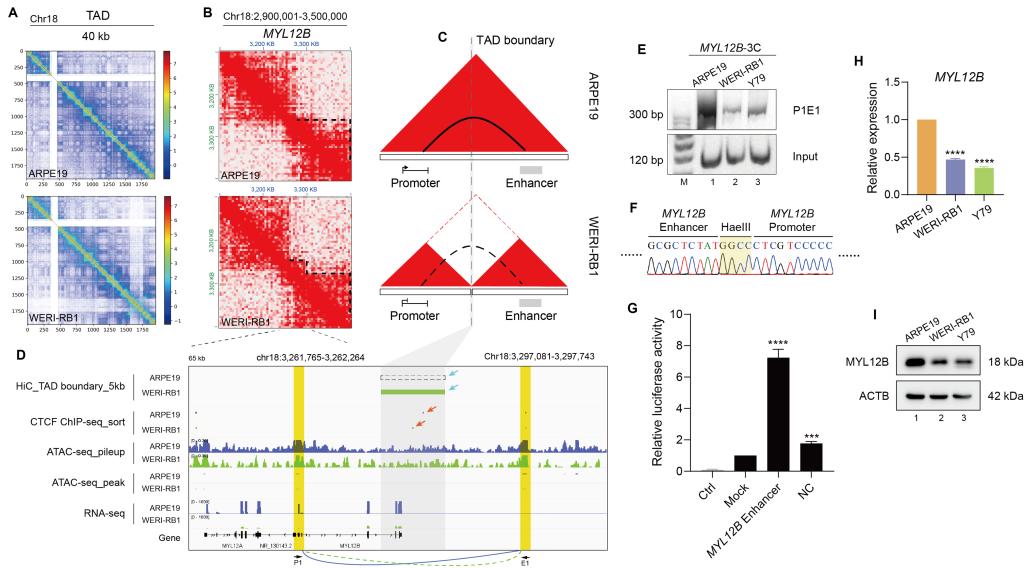
此外💪🏼,與ARPE19細胞相比👨🍼🧓🏼,WERI-RB1細胞12號染色體上的促癌基因CYP27B1位點發生TAD融合現象,使得CYP27B1的啟動子(P2)和增強子(E2)共存於同一個TAD中,形成特異性染色質環💳,導致腫瘤細胞中CYP27B1轉錄上調。
綜上所述🧖🏼♀️,該方法及其識別的靶點轉錄機製,為進一步理解TAD重構在癌基因和抑癌基因轉錄調控中的作用提供了新的見解,有望為從染色質三維構象重塑角度發現惡性腫瘤治療新靶點提供重要線索。
恒达平台丁天宜、傅沙镠與張曉宇為論文共同第一作者🦶,恒达平台劉琦⛱、張赫為論文共同通訊作者🛹。同時,作為恒达平台對口支援井岡山大學的聯合學術成果,該研究工作獲得了恒达平台生命科學與技術學院、恒达平台附屬東方醫院、江西省器官發育與表觀遺傳重點實驗室、井岡山大學附屬醫院臨床醫學研究中心👩🏿🏭、井岡山大學醫學部和生命科學學院的大力支持,受到了國家重點研發計劃🚵🏼💎、國家自然科學基金委和上海市科委等項目的資助。
論文鏈接🎧:https://academic.oup.com/gpb/advance-article/doi/10.1093/gpbjnl/qzae034/7667293